1. EasyMultiProfiler
EasyMultiProfiler包采用了面向对象的编程方法,集合了多个数据分析模块,实现了数据在不同模块之间的流畅传递,从而使得整体分析流程标准化和清晰化。EasyMultiProfiler不仅包括当前主流的数据预处理方法,还具备强大的分析能力,不仅涵盖多种主流的分析方法(包括:多样性分析、聚类分析、相关性分析、差异性分析和降维分析等),还支持GSEA富集分析、WGCNA分析以及多组学项目的联合分析。用户可以利用这些功能快速探索数据特征和模式,并通过多种可视化方式(包括:箱型图、散点图、富集点状图、富集网络图、双变量图、富集曲线图、热图、桑基图和结构柱状图等)直观地解释分析结果。总而言之,EasyMultiProfiler为用户提供了一站式的数据处理、分析和可视化服务,简化了大量的中间过程,且逻辑清晰,对于初学者也是非常友好的。
1.1 EasyMultiProfiler的安装
R的版本必须在4.3.3及以上。 (重要!!!)
1.1.1 首次安装
(1) Mac os 用户:
安装方法① (推荐)
第一步: 安装gfortran编译环境。
R中有很多包在安装时需要编译环境。为了确保最大的兼容性,建议新用户安装以下版本,而非最新版本的gfortran。建议安装后重启电脑。
下载地址:官方原始地址
第二步: 使用pak进行安装
if (!requireNamespace("pak", quietly=TRUE)) install.packages("pak")
pak::pak("liubingdong/EasyMultiProfiler")
library(EasyMultiProfiler)
安装方法②
第一步: 安装clusterProfiler
if (!requireNamespace("BiocManager", quietly=TRUE)) install.packages("BiocManager")
BiocManager::install("clusterProfiler")
library(clusterProfiler)
第二步: 使用remotes进行安装
if (!requireNamespace("remotes", quietly=TRUE)) install.packages("remotes")
remotes::install_github("liubingdong/EasyMultiProfiler")
library(EasyMultiProfiler)
(2) Windows用户:
第一步: 安装RTool。
根据已安装的R版本,安装对应版本的RTool(例如:R 4.3.x 需安装 RTool4.3,R 4.4.x 需安装 RTool4.4)。
- RTool安装步骤:在下载页面,依此点击
[Download R for Windows]→[Rtools])
第二步:成功安装对应版本的RTool之后,建议重启电脑。
第三步:使用pak包进行安装。
if (!requireNamespace("pak", quietly=TRUE)) install.packages("pak")
pak::pak("liubingdong/EasyMultiProfiler")
library(EasyMultiProfiler)
1.1.2 更新版本
EasyMultiProfiler包在持续更新更多的分析和可视化模块,当需要更新版本时,可以再次运行pak即可进行更新。
pak::pak("liubingdong/EasyMultiProfiler")
library(EasyMultiProfiler)
1.2 EasyMultiProfiler快速开始
为了快速熟悉EasyMultiProfiler的功能和强大之处,我们可以先从各种有趣的示例开始:1.3 EasyMultiProfiler基本知识
1.3.1 EasyMultiProfiler的数据对象容器
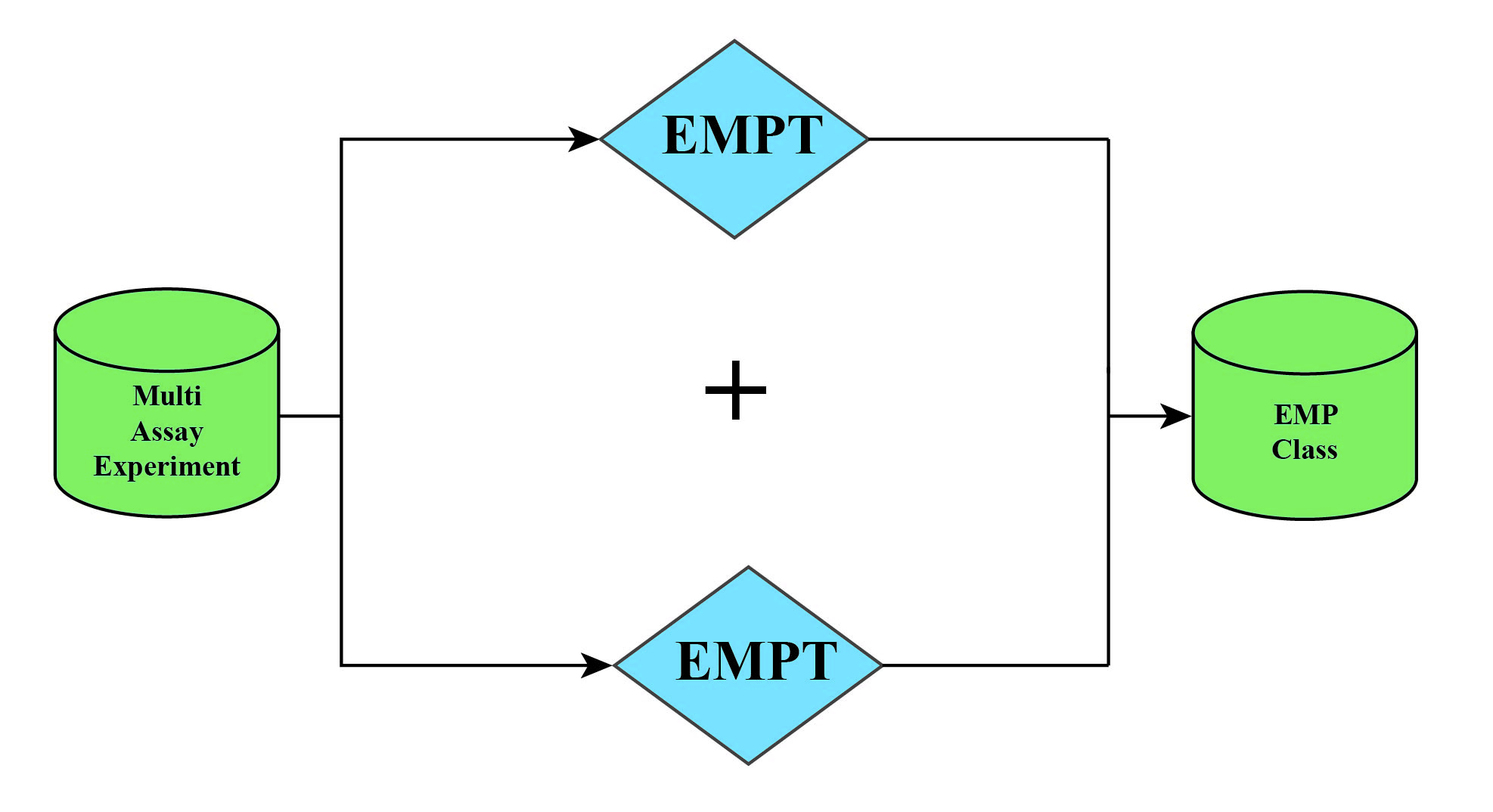
MAE对象:即MultiAssayExperiment。MAE对象是用户需要初始创建的对象,通常是由多个组学项目的实验数据(assay)和与之对应的样本相关数据(coldata)、特征相关数据(rowdata)组成(相关定义详见本章节1.2.2),也支持创建单一组学项目的MAE对象。EMPT对象:即EMP-transporter。从MAE对象中提取出单一组学项目,即形成EMPT对象。EMPT对象继承SummarizedExperiment数据存储对象,主要包含assay、rowdata、coldata和各种流程分析所需参数等。EMP对象:当多个EMPT对象进行联合分析时,可以将这些EMPT对象合并成一个EMP对象。
①为了尽可能保证原始数据不会被篡改,数据分析过程中从
MAE→EMPT的过程是单向的,即不能将EMPT对象重新放回MAE对象。②如需修改
MAE对象内的EMPT对象,请直接调整EMPT对象,重新组装成MAE对象。③当进行联合分析时,可以直接使用
+符号将EMPT对象组合成EMP对象,从而进行组学联合分析流程。④当参与合并的组学项目名称一致时,将会自动修改后一个组学项目的名称,以确保
EMPT对象中组学项目名称的唯一性。
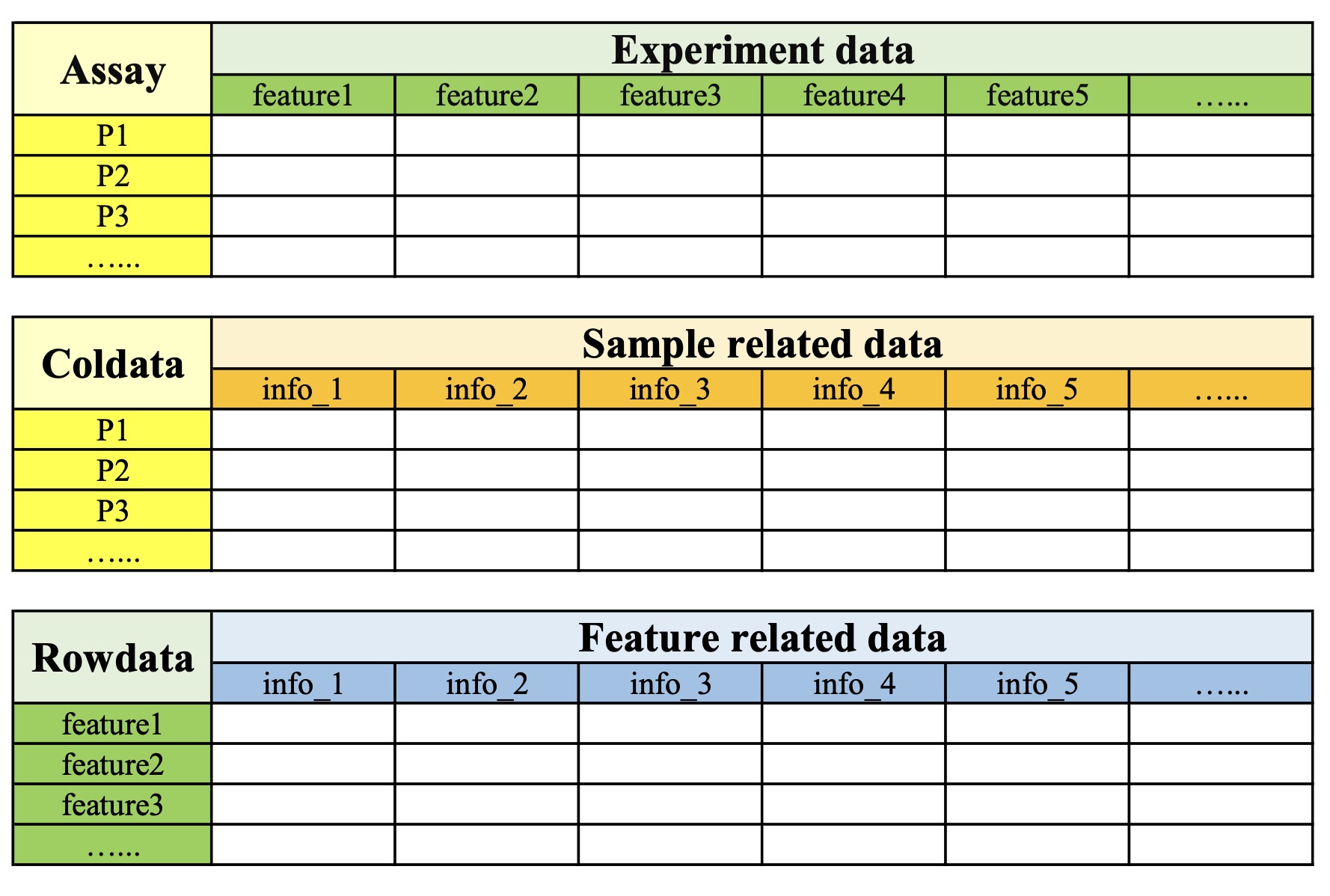
1.3.2 EasyMultiProfiler的数据组成
EasyMultiProfiler对象内存储的每个组学项目的数据均包含以下三项内容:assay、coldata 、rowdata。assay是指组学项目的实验数据;coldata是指样本相关数据;rowdata是指特征相关数据。
| Assay | Coldata | Rowdata | |
|---|---|---|---|
| Column | Feature | Sample related data | Feature related data |
| Row | Sample ID | Sample ID | Feature |
1.3.3 EasyMultiProfiler的数据展现
EasyMultiProfiler包采用tibble格式(一种数据框类型)来呈现数据,在tibble的列名下,用户可以查看对应列的数据类型。常见的数据类型包括:int-整数型、dbl-浮点型、chr-字符串型、lgl-逻辑型(True和False)、fct-因子型、date-日期型、dttm-日期时间型。
1.3.4 EasyMultiProfiler的数据传递
EasyMultiProfiler包使用|>操作符以帮助数据传递,即将前一个模块的输出作为后一个模块的输入(与Magrittr包的%>%操作符功能相同),从而实现连续的数据处理操作。由于数据流程中传递的是EMPT对象,因此整个数据流程几乎没有强制的分析顺序。用户可以自由地在各个模块中进行数据分析探索,而无需担心数据传递的匹配问题。
1.3.5 EasyMultiProfiler的行动参数
EasyMultiProfiler包进行数据分析时,组学项目数据将被存储在EMPT对象内,并在功能模块之间进行传递。在用户完成某个功能模块的分析后,可以使用参数action提取该模块产生的原始分析结果。此参数默认值为action='add',代表当前功能模块的分析结果将会被添加到EMPT对象内进行传递;action='get',代表当前功能模块的分析结果将直接输出。
模块
EMP_filter中的参数action用于指定此模块操作是剔除(kick)或者保留(select)。
1.3.6 EasyMultiProfiler的缓存机制
EasyMultiProfiler包的主要分析模块均具备缓存快照机制,即:当重复使用相同的数据进行分析时,分析流程将自动根据哈希值从缓存中提取对应的数据分析结果,从而避免重复计算。这种缓存特性极大地提升了分析流程的便利性,避免了在传统的数据分析中为了存储复杂运算结果而创建多个中间变量的麻烦。用户只需通过|>操作符传递当前的计算结果,即可顺利完成每一个分析步骤,使得整个分析流程更加清晰和简洁。
① 缓存机制考虑了数据、参数和模块体的差异,基本消除了可能的数据冲突问题。
② 如果需要重新计算,可以使用
use_cached=FALSE取消本次模块的缓存。
data(MAE)
## First run
system.time({
MAE |>
EMP_assay_extract('geno_ec') |>
EMP_WGCNA_cluster_analysis(RsquaredCut = 0.85)
})
## Second run
system.time({
MAE |>
EMP_assay_extract('geno_ec') |>
EMP_WGCNA_cluster_analysis(RsquaredCut = 0.85)
})
## Close the cached
system.time({
MAE |>
EMP_assay_extract('geno_ec') |>
EMP_WGCNA_cluster_analysis(RsquaredCut = 0.85,use_cached = FALSE)
})
